Table des matières
- Définition
- I. La méthode RFLP
- II. L’analyse des SNP par puce à ADN
- III. Applications à la sécurité alimentaire
Définition
Entre les espèces très proches ou au sein d’une espèce, les individus peuvent présenter d’infimes variations dans leur génome qui :
- Modifient la séquence d’une protéine : remplacement d’un acide aminé, perte ou ajout d’acides aminés. Cette modification altère ou non le fonctionnement de la protéine ;
- Modifie la physiologie des cellules : par altération de séquences régulatrices de l’expression du génome ;
- Ne modifient en rien le fonctionnement de la cellule, si elles interviennent dans des régions du génome considérées comme non-codante et non-régulatrices.
Ces variations peuvent avoir plusieurs échelles :
- Un seul nucléotide varie : on parle de polymorphisme d’un nucléotide unique, ou SNP (single nucleotid polymorphism). On estime qu’il existe 10 x 106 de SNP chez l’humain, soit un tout les 300 pb ;
- Ajout/retrait d’une série de nucléotides (de 1 base à 10 kb) par rapport à une séquence de référence. On compte 400 000 exemples de ces Indel (insertion/délétion) chez l’humain ;
- Les répétitions de séquences ou SSR (simple sequence repeat) : on les appelle également les microsatellites. Il s’agit d’une séquence de deux à quelques nucléotides, qui sont répétées plusieurs fois. Exemple : 14 répétitions de la séquence ATC sera noté (ATC)14. D’un individu à un autre, le nombre de répétitions peut être très variable. Il existe environ deux millions de microsatellites dans le génome humain ;
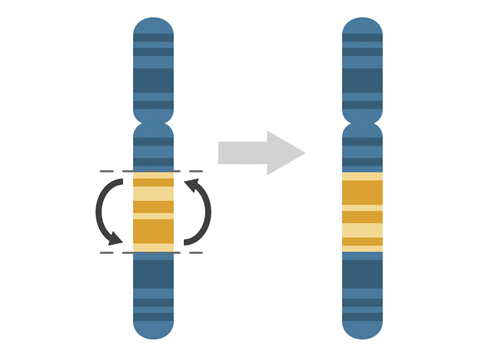
- Inversion : retournement de la séquence en base au sein d’un chromosome. La taille du retournement couvre jusqu’à plusieurs milliers de bases. C’est un évènement rare, il n’existe pas plus de 500 évènements dans le génome humain.
Nous allons voir comment le génotypage peut permettre d’identifier des microorganismes, ou garantir la traçabilité des aliments (la viande notamment).
I. La méthode RFLP
I.A. Principe de la méthode
Le RFLP (= restriction fragment length polymorphism ou polymorphisme de la longueur des fragments de restriction) est une méthode qui permet de mettre en évidence des variations du génome à l’échelle de quelques centaines de bases, principalement les SNP et les indels.
Rappel : une enzyme de restriction est une enzyme qui coupe l’ADN au niveau d’une séquence en bases bien spécifique, qui est palindromique (symétrique).
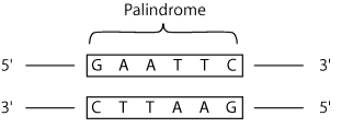
Cette séquence est spécifique à l’enzyme de restriction. Généralement, les enzymes de restriction possèdent leur propre séquence de coupure.
Lorsqu’un échantillon d’ADN est soumis à un traitement par une enzyme de restriction, il sera coupé en autant de fragments qu’il y a de séquences de coupures (+ 1).
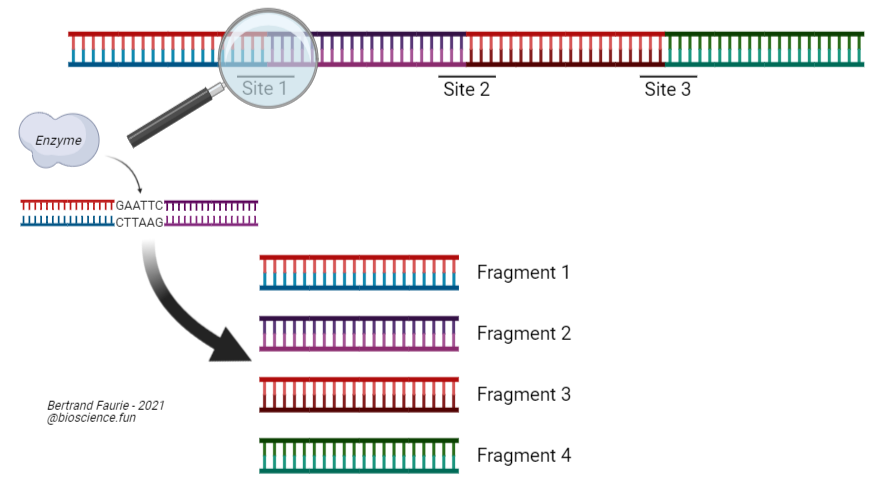
Les fragments sont ensuite séparés sur gel d’agarose ou d’acrylamide et révélés soit par une sonde spécifique marquée, soit par incorporation de nucléotides marqués lors de la synthèse de l’échantillon d’ADN, par PCR notamment. Les fragments se séparent en fonction de leur taille respective, les plus petits migrant le plus loin.
Dans le cas de la figure, tous les fragments font la même taille, il n’y aura donc qu’une seule bande sur le gel.
Comment ce traitement peut-il mettre en évidence une variation du génome telles qu’elles sont décrites dans « définition » ?
I.B. Mise en évidence d’un SNP
I.C. Mise en évidence d’un indel
Travail à faire en classe. En utilisant le document 03 et le profil de migration des fragments obtenus, imaginer l’altération du profil de migration dans les cas suivants :
- SNP au niveau du site de restriction 2
- SNP en dehors d’un site de restriction
- Perte de la moitié de la séquence comprise entre les sites 2 et 3
- Doublement de la séquence entre les sites 1 et 2
II. L’analyse des SNP par puce à ADN
II.A. Principe de la méthode
Quelques éléments pour bien comprendre la vidéo !
- Il s’agit d’une méthode parmi d’autres de détermination des SNP ;
- La méthode est dérivée de la méthode de séquençage développée par Illumina (voir le cours sur le séquençage) ;
- Pour chaque SNP, il existe deux allèles : un paternel et l’autre maternel. Le SNP peut être identique pour les deux allèles ou différent. Dans l’exemple, il s’agit d’un SNP qui peut adopter une configuration G ou A. La personne peut donc être AA, AG ou GG.
- La sonde qui permet de capturer la séquence contenant le SNP fait environ 50 pb de long, et se situe en amont de la position du SNP. Elle s’arrête précisément au nucléotide précédant la position du SNP. De telle sorte que lors de la phase de synthèse (méthode Illumina), le nucléotide terminateur marqué qui sera ajouté (ddNTP) sera forcément le complémentaire de la base constituant le SNP !
II.B. La puce à ADN
La puce à ADN (micro- ou macroarray) est une collection de fragments d’ADN synthétisés et contrôlés (des sondes), fixés sur une surface en verre. Il existe des adaptations, comme dans le cas de la technique Illumina de détermination des SNP pour laquelle les sondes sont fixées à des billes de silicone, elles-mêmes enchâssées dans un nano-puit creusé dans la lame en verre.
Les sondes déposées sur la lame dépendent du type d’information qui est recherché :
- Des gènes qui s’expriment ?
- Des séquences spécifiques ?
- Des modifications de la séquence ?
- Des SNPs ?
- …
Elles dépendent également du type d’organisme qui est étudié : il existe des sondes pour tous les organismes séquencés.
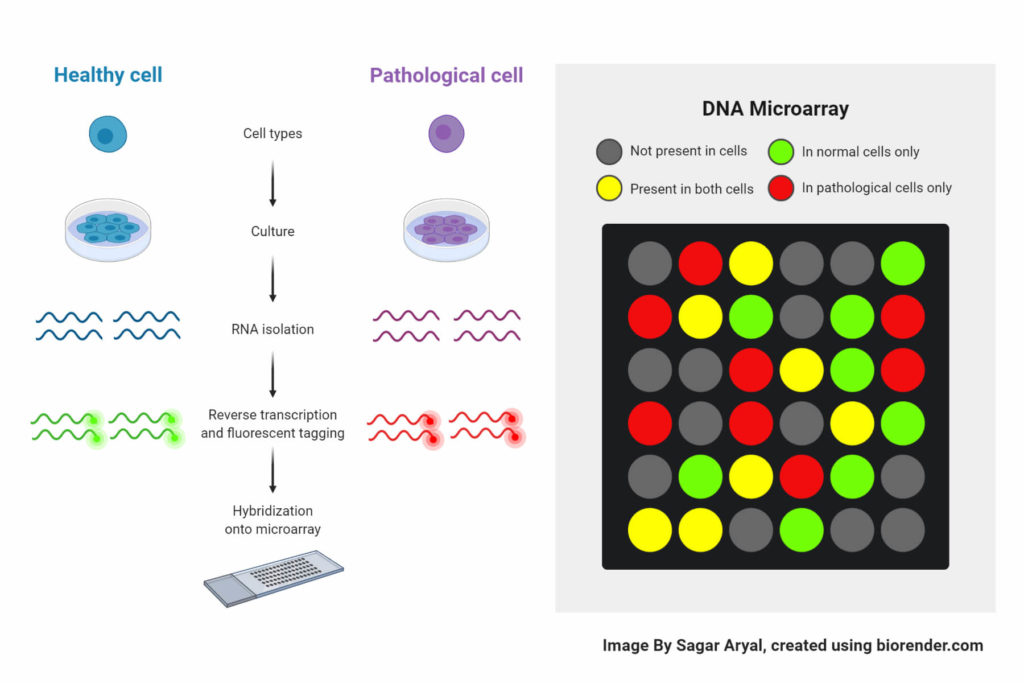
Le protocole pour mettre en oeuvre une puce à ADN va dépendre du matériel de départ (ADN génomique, ARNm, amplicons) et du type d’information recherché. Mais, le résultat sera quasiment toujours le même :
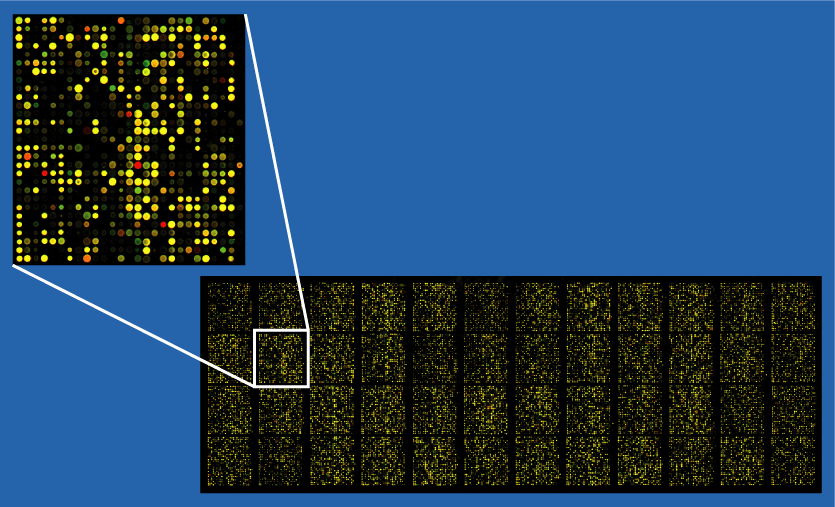
Chaque point correspond à une sonde se rapportant à une information génétique. Les couleurs proviennent des marqueurs utilisés pour visualiser les fragments d’ADN qui se sont hybridés avec les sondes. On teste deux conditions, chacune associée à une des couleurs : si la condition A est marqué en vert et la condition B, en rouge, alors :
- Plot vert : A majoritaire ;
- Plot rouge : B majoritaire ;
- Plot jaune : A et B équivalents.
Il existe sur les puces des contrôles qualités, notamment sous forme de sonde se rapportant à des informations génétiques constantes : le résultat attendu est jaune.
Exemple de l’utilisation des puces à ADN dans le cadre d’une analyse des profils d’expression génétique dans des cellules saines et des cellules pathologiques (cancer/parasitose/…).
III. Applications à la sécurité alimentaire
Identification des souches bactériennes.
Traçabilité des lots de viande
Traçabilité des lots de viande 02
Traçabilité des eaux d’hydrofracturation dans la prospection des produits pétroliers
Biomarquage des espèces végétales et produits dérivés pour garantir la traçabilité des produits
